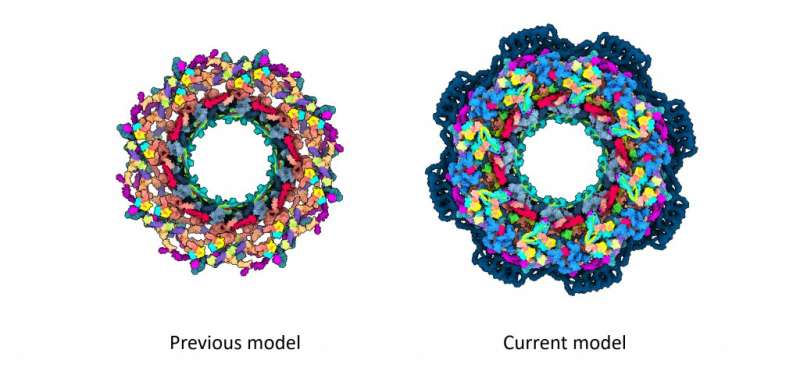
Sammenligning av modellene av menneskelig kjernefysisk porekompleks oppnådd før (venstre) og etter bruk av AlphaFold2 kombinert med cryo-ET (høyre). Det menneskelige kjernefysiske porekomplekset har en totalvekt på 120 MDa. De tidligere modellene dekket bare opptil 35 MDa. Den nye modellen dekker 70 MDa. Kreditt: Agnieszka Obarska-Kosińska/EMBL og MPI of Biophysics
Kombinasjonen av AlphaFold2 med eksperimentelle og beregningstekniske teknikker har hjulpet forskere med å finne ut arkitekturen til det menneskelige kjernefysiske porekomplekset i større detalj enn noen gang før.
Det menneskelige kjerneporekomplekset (NPC) er en ekte molekylær gigant, som sitter på membranen som skiller kjernen fra cytoplasmaet. Den er smultringformet og fungerer som både en gateway og et sjekkpunkt for molekyler som beveger seg mellom cytoplasma og kjernen. Derved letter NPC grunnleggende prosesser i cellen, slik som genuttrykk og translasjon. Det kjernefysiske transportsystemet spiller også en rolle i flere sykdommer, inkludert nevrodegenerative lidelser, kreft og virusinfeksjoner.
Hva er strukturen til NPC? Hvordan limes proteinene sammen? Hvordan fester den seg til kjernemembranen? Disse og andre spørsmål har nå blitt besvart av Kosinski Group ved EMBL Hamburg og Center for Structural Systems Biology (CSSB), Beck og Hummer Labs ved Max Planck Institute of Biophysics, og samarbeidspartnere. De skapte den mest komplette modellen av den menneskelige NPC til dags dato ved å kombinere proteinstrukturprediksjonsprogrammet AlphaFold2 med teknikker som kryo-elektrontomografi, enkeltpartikkel-kryo-EM og integrativ modellering.
For strukturbiologer er den menneskelige NPC et utfordrende, men spennende 3D-puslespill, med rundt 30 forskjellige proteiner hver til stede i flere kopier. Dette utgjør rundt 1000 puslespillbrikker, som danner en rund kjerne med omkringliggende fleksible deler. Til nå har de mest nøyaktige modellene av den menneskelige NPC-kjernen bare dekket 46% av strukturen. Men nå, basert på to tiår med tidligere forskning på feltet, har forskere laget en ny modell av NPC-strukturen som dekker mer enn 90% av kjernen.
Mens tidligere foreslåtte NPC-modeller hadde hull og inneholdt noen proteiner bare i fragmenter, fjerner den nye modellen mye av denne tvetydigheten.
“Det er som når du demonterer og setter sammen en elektronisk enhet. Det vil alltid være noen skruer igjen, og du vet bare ikke hvor de skal være,” sa EMBL-gruppeleder Jan Kosinski, som er medleder i etterforskningen. “Vi klarte endelig å passe de fleste av dem, og nå vet vi nøyaktig hvor de er, hva de gjør og hvordan.”
Eksperimentering og kunstig intelligens fungerer sammen
Hvordan oppnådde forskerne dette? Nøkkelen var å kombinere flere eksperimentelle og beregningsmetoder. Dette gjorde det mulig for forskerne å visualisere NPC i forskjellige skalaer og detaljnivåer.
For eksempel, for å modellere den generelle silhuetten til NPC, brukte forskerne kryo-elektrontomografi. Med denne teknikken var de i stand til å observere NPC i dets cellulære miljø, i stedet for isolert. Flere detaljer om de individuelle proteinbyggesteinene ble avslørt av AlphaFold2, et kunstig intelligensbasert program som forutsier protein strukturer, laget av selskapet DeepMind.
“AlphaFold2 var et banebrytende øyeblikk for oss,” sa Agnieszka Obarska-Kosińska, postdoc som utførte den molekylære modelleringen. “Før kjente vi ikke strukturen til mange proteiner i NPC. Du kan ikke sette sammen et puslespill når du ikke vet hvordan brikkene ser ut. Men AlphaFold2 kombinert med andre tilnærminger gjorde det mulig for oss å forutsi disse formene.”
For å avgrense bildet ytterligere brukte forskerne ColabFold, en versjon av AlphaFold2 modifisert av det vitenskapelige samfunnet for å modellere interaksjoner mellom proteiner. Dette tillot dem å visualisere hvordan de forskjellige puslespillbrikkene kombineres for å danne mindre underkomplekser, og hvordan disse underkompleksene deretter limes sammen for å danne NPC.
Til slutt satte de alle bitene sammen ved hjelp av programvaren Assembline tidligere utviklet av Kosinski Group, og validerte den mot eksperimentelle data.
Den resulterende modellen var så komplett og detaljert at den gjorde det mulig for forskerne å lage tidsløste molekylære simuleringer som forklarer hvordan NPC-proteinene og kjernemembranen samhandler for å skape en stabil pore og hvordan den reagerer på mekaniske signaler.
Fremtidige retninger
Dette arbeidet var et stort sprang fremover for NPC-forskning, men det er fortsatt mye igjen å utforske.
“Dette arbeidet eksemplifiserer hvordan, i fremtiden, strukturell biologi vil omfavne cellebiologi for å lage atommodeller av stadig større samlinger av molekyler som utfører forskjellige funksjoner i forskjellige deler av cellen,” sa Martin Beck. Gerhard Hummer er enig: “Vi kan nå tenke på å bygge en komplett dynamisk modell av NPC og simulere atomtransport i atomdetaljer.”
Kosinski Group vil sikte sitt fremtidige arbeid på å utvikle automatiske metoder for integrering av struktur- og mikroskopidata ved hjelp av AlphaFold2 og deres egen programvare Assembline. De planlegger å bruke disse tilnærmingene til å studere molekylære prosesser som driver virusinfeksjoner.
Forskningen ble publisert i Vitenskap.
Observer det hemmelige livet til molekyler inne i cellen
Shyamal Mosalaganti et al, AI-basert strukturprediksjon styrker integrativ strukturell analyse av menneskelige kjernefysiske porer, Vitenskap (2022). DOI: 10.1126/science.abm9506
Levert av
European Molecular Biology Laboratory
Sitering: Forvirring av strukturen til en molekylær gigant (2022, 10. juni) hentet 11. juni 2022 fra https://phys.org/news/2022-06-puzzling-molecular-giant.html
Dette dokumentet er underlagt opphavsrett. Bortsett fra enhver rettferdig handel med formålet med private studier eller forskning, kan ingen del reproduseres uten skriftlig tillatelse. Innholdet er kun gitt for informasjonsformål.