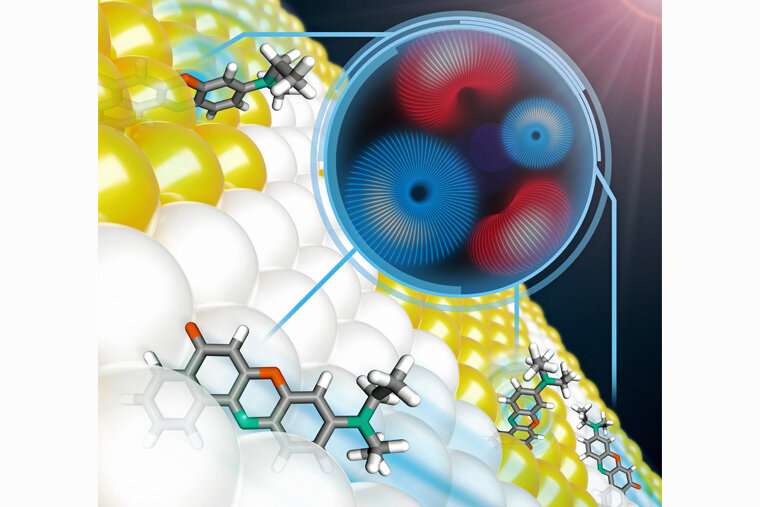
“Molekyler samhandler alltid med omgivelsene sine. Det har vært en kamp for enhver teknologi å forstå denne interaksjonen,” sa Matthew Lew, assisterende professor ved McKelvey School of Engineering. Den kampen kan bli litt lettere takket være den siste forskningen fra laboratoriet hans, som åpner for en ny verden av dynamisk bildebehandling. Kreditt: Oumeng Zhang
Forskning fra laboratoriet til Matthew Lew ved Washington University i St. Louis tilbyr helt nye måter å se de helt små.
Forskningen – to artikler av Ph.D. studenter ved McKelvey School of Engineering – ble publisert i tidsskriftene OPTISK og Nanobokstaver.
De har utviklet ny maskinvare og algoritmer som lar dem visualisere byggesteinene i den biologiske verden utenfor tre dimensjoner på en måte som til nå ikke var gjennomførbar. Tross alt, celler er 3D-objekter og fulle av «ting» – molekyler – som beveger seg rundt, roterer, snurrer og tumler for å drive selve livet.
Som tradisjonelle mikroskoper er arbeidet til to Ph.D. studenter i Lew-laben, Tingting Wu og Oumeng Zhang, bruker lys å kikke inn i den mikroskopiske verden – men deres innovasjoner er alt annet enn tradisjonelle. For øyeblikket, når folk bruker lys i bildebehandling, er de sannsynligvis interessert i hvor sterkt lyset er eller hvilken farge det er. Men lys har andre egenskaper, inkludert polarisering.
“Oumengs arbeid vrir polarisasjonen av lys,” sa Lew, assisterende professor ved Preston M. Green Department of Electrical & Systems Engineering. “På denne måten kan du både se hvordan ting oversetter seg (beveger seg i rette linjer) og roterer samtidig” – noe tradisjonell bildebehandling ikke gjør.
“Utviklingen av ny teknologi og evnen til å se ting vi tidligere ikke kunne se er spennende,” sa Zhang. Denne unike evnen til å spore både rotasjon og posisjon samtidig gir ham unik innsikt i hvordan biologiske materialer—menneskelige celler og patogener, for eksempel – samhandler.
Wus forskning gir også en ny måte å avbilde cellemembraner og på en måte se innsiden av dem. Ved hjelp av fluorescerende spormolekyler kartlegger hun hvordan sporstoffene interagerer med fett- og kolesterolmolekyler i membranen, og bestemmer hvordan lipidene er ordnet og organisert.
“Noen cellemembran, enhver kjerne, alt i cellen er en 3D-struktur,” sa hun. “Dette hjelper oss å undersøke hele bildet av et biologisk system. Dette gjør at vi, for enhver biologisk prøve, kan se utover tre dimensjoner – vi ser 3D-strukturen pluss tre dimensjoner av molekylær orientering, noe som gir oss 6D-bilder.”
Forskerne utviklet databehandlingsteknologi, som synergerer programvare og maskinvare sammen, for å lykkes med å se det tidligere usynlige.
“Det er en del av innovasjonen,” sa Lew. “Tradisjonelt har biologiske avbildningslaboratorier vært bundet til hva kommersielle produsenter lager. Men hvis vi konstruerer ting annerledes, kan vi gjøre så mye mer.”
Vortex-mikroskop ser mer enn noen gang før
Tingting Wu et al, Dipole-spread-function engineering for samtidig måling av 3D-orienteringer og 3D-posisjoner til fluorescerende molekyler, OPTISK (2022). DOI: 10.1364 / OPTICA.451899
Oumeng Zhang et al, løser den tredimensjonale rotasjons- og translasjonsdynamikken til enkeltmolekyler ved bruk av radielt og azimutalt polarisert fluorescens, Nanobokstaver (2022). DOI: 10.1021/acs.nanolett.1c03948
Levert av
Washington University i St. Louis
Sitering: Nytt lys skinner på cellemembraner med forbedret bildebehandling (2022, 23. mai) hentet 24. mai 2022 fra https://phys.org/news/2022-05-cell-membranes-imaging.html
Dette dokumentet er underlagt opphavsrett. Bortsett fra enhver rettferdig handel med formålet med private studier eller forskning, kan ingen del reproduseres uten skriftlig tillatelse. Innholdet er kun gitt for informasjonsformål.